مدل سازی انتقال دو بعدی با MODFLOW، MT3D-USGS و Flopy

ساخت مدل های هیدروژئولوژیکی می تواند وقت زیادی برای ساخت و ساز، تجسم نتایج و کالیبراسیون را استفاده کند. مهم است که از ابزارهایی استفاده کنید که می توانند این کارها را بهینه سازی کنند و در زمان صرفه جویی کرده و در تجزیه و تحلیل سیستم آن را به کار گیرید.
در این فرصت ما Flopy را برای تکرار یک مدل حمل و نقل 2D از یک پست قبلی استفاده خواهیم کرد با مجموعه ای متنوع از اسکریپت های پایتون است که می تواند برای اجرای MODFLOW و MT3D، از جمله برنامه های زیرزمینی مرتبط با MODFLOW، به روش ساده و کارآمد مورد استفاده قرار گیرد. به نظر می رسد که این ابزار به طور خودکار فرآیند ایجاد مدل های آب زیرزمینی را مفید می داند، زیرا تغییرات شرایط مرزی فقط با تغییر دادن فایل متنی انجام می شود.
علاوه بر این، MT3D-USGS برای مدل سازی حمل و نقل مورد استفاده قرار خواهد گرفت. این یک نسخه به روز شده از کد منبع حمل و نقل زیرزمینی MT3DMS است که قابلیت های جدید مدل سازی حمل و نقل را فراهم می کند که انعطاف پذیری بیشتری در شبیه سازی حمل و نقل حلال و حمل و نقل حلال واکنش را نشان می دهد.

برای یافتن تمامی مطالب مرتبط با این مطلب در سایت از جستجوی سایت در حاشیه سمت راست و بالای صفجه استفاده فرمایید.

ورود به بخش آموزش های متنی GMS


دانلود آخرین نسخه نرم افزار GMS

دریافت لایسنس ارزیابی (14 روزه)

برای سفارش انجام مدل سازی اینجا کلیک کنید
کد ورودی
این کد کامل برای مدل آبهای زیرزمینی در Flopy است:
شما می توانید این کد را به عنوان یک نوت بوک Jupyter در اینجا دانلود کنید.
#Import
%matplotlib inline
import numpy as np
import matplotlib.pyplot as plt
import flopy
import flopy.modflow as mf
import flopy.mt3d as mt
import flopy.utils as fu#MODFLOW 2005
modelname = 'example'
mf_model = mf.Modflow(modelname = modelname, exe_name='mf2005.exe')#DIS file
Lx = 310
Ly = 310
nrow = 31
ncol = 31
nlay = 1
delr = Lx / ncol
delc = Ly / nrow
top = np.ones((nrow, ncol))
botm = np.zeros((nrow, ncol))
perlen = 27
dis = mf.ModflowDis(mf_model, nlay, nrow, ncol, delr = delr, delc = delc,
top = top, botm = botm, laycbd = 0, itmuni=4, perlen = perlen,
nstp = 1)# Output Control: Create a flopy output control object
oc = mf.ModflowOc(mf_model)#BCF file
laycon=0 #confined
tran=1.0 #transmissivity
bcf = flopy.modflow.mfbcf.ModflowBcf(mf_model,laycon=0, tran=1.0)#BAS file
ibound = np.ones((nlay, nrow, ncol)) #active
ibound[0, 0, :31] = -1 #constant head
ibound[0, 30, :31] = -1
ibound[0, :31, 0] = -1
ibound[0, :31, 30] = -1
strt=15 #starting head
iboundbas = mf.ModflowBas(mf_model, ibound = ibound, strt = strt)#PCG file
pcg = flopy.modflow.mfpcg.ModflowPcg(mf_model, mxiter=20, iter1=30, hclose=1e-03, rclose=1e-03, relax=1.0)#CHD
#[lay, row, col, shead, ehead]
chd=15
chd_data = []
for c in range(30):
dd = np.array([0, 0, c, chd, chd])
chd_data.append(dd)
for c in range(31):
dd = np.array([0, c, 30, chd, chd])
chd_data.append(dd)
for c in range(30):
dd = np.array([0, 30, c, chd, chd])
chd_data.append(dd)
for c in range(1,30):
dd = np.array([0, c, 0, chd, chd])
chd_data.append(dd)
stress_period_data = {0:chd_data}
stress_period_datachd = mf.mfchd.ModflowChd(mf_model, stress_period_data=stress_period_data)#WELL
#[lay, row, col, pumping rate]
pumping_rate = 100 #m3/d
wel_sp1 = [[0, 15, 15, pumping_rate]]
stress_period_data = {0: wel_sp1}
wel = flopy.modflow.ModflowWel(mf_model, stress_period_data=stress_period_data)#LMT Linkage with MT3DMS for multi-species mass transport modeling
lmt = flopy.modflow.ModflowLmt(mf_model, output_file_name='mt3d_link.ftl')#Write input files
mf_model.write_input()
# run the model
mf_model.run_model()#Plot model results
import matplotlib.pyplot as plt
import flopy.utils.binaryfile as bf
# Create the headfile object
headobj = bf.HeadFile(modelname+'.hds')
head = headobj.get_data(totim=27)
times = headobj.get_times()
# Setup contour parameters
levels = np.arange(0, 90, 5)
extent = (delr/2., Lx - delr/2., delc/2., Ly - delc/2.)
# Make the plots
plt.subplot(1, 1, 1, aspect='equal')
plt.title('Head distribution (m)')
plt.imshow(head[0, :, :], extent=extent, cmap='YlGnBu', vmin=0., vmax=90.)
plt.colorbar()
contours = plt.contour(np.flipud(head[0, :, :]), levels=levels, extent=extent, zorder=10)
plt.clabel(contours, inline=1, fontsize=10, fmt='%d', zorder=11)
plt.show()#MT3D-USGS
namemt3d='modelnamemt3d'
mt_model = mt.Mt3dms(modelname=namemt3d, version='mt3d-usgs', exe_name='MT3D-USGS_64.exe', modflowmodel=mf_model)#BTN file
icbund = np.ones((nlay, nrow, ncol))
icbund[0, 15, 15] = -1 #constant concentration
btn = flopy.mt3d.Mt3dBtn(mt_model, sconc=0.0, prsity=0.3, thkmin=0.01, munit='g', icbund=icbund)#ADV file
mixelm = -1 #Third-order TVD scheme (ULTIMATE)
percel = 1 #Courant number PERCEL is also a stability constraint
adv = flopy.mt3d.Mt3dAdv(mt_model, mixelm=mixelm, percel=percel)#GCG file
mxiter = 1 #Maximum number of outer iterations
iter1 = 200 #Maximum number of inner iterations
isolve = 3 #Preconditioner = Modified Incomplete Cholesky
gcg = flopy.mt3d.Mt3dGcg(mt_model, mxiter=mxiter, iter1=iter1, isolve=isolve)#DSP file
al = 10 #longitudinal dispersivity
dmcoef = 0 #effective molecular diffusion coefficient
trpt = 0.1 #ratio of the horizontal transverse dispersivity to the longitudinal dispersivity
trpv = 0.01 #ratio of the vertical transverse dispersivity to the longitudinal dispersivity
dsp = mt.Mt3dDsp(mt_model, al=al, dmcoef=dmcoef, trpt=trpt, trpv=trpv)#SSM file
itype = flopy.mt3d.Mt3dSsm.itype_dict()
#[K,I,J,CSS,iSSType] = layer, row, column, source concentration, type of sink/source: well-constant concentration cell
ssm_data = {}
ssm_data[0] = [(0, 15, 15, 1.0, 2)]
ssm_data[0].append((0, 15, 15, 1.0, -1))
ssm = flopy.mt3d.Mt3dSsm(mt_model, stress_period_data=ssm_data)#Write model input
mt_model.write_input()
#Run the model
mt_model.run_model()#Plot concentration results
conc = fu.UcnFile('MT3D001.UCN')
conc.plot(totim=times[-1], colorbar='Concentration (mg/l)', cmap='Blues')
plt.title('Concentration distribution (mg/l)')
plt.show()
مدیر سایت: بهزاد سرهادی کارشناس ارشد مهندسی آب
شناسه تلگرام مدیر سایت: SubBasin@
نشانی ایمیل: behzadsarhadi@gmail.com
(سوالات تخصصی را در گروه تلگرام ارسال کنید)
_______________________________________________________
پروژه تخصصی در لینکدین




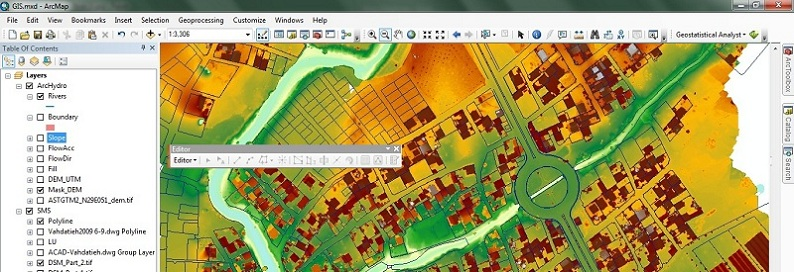
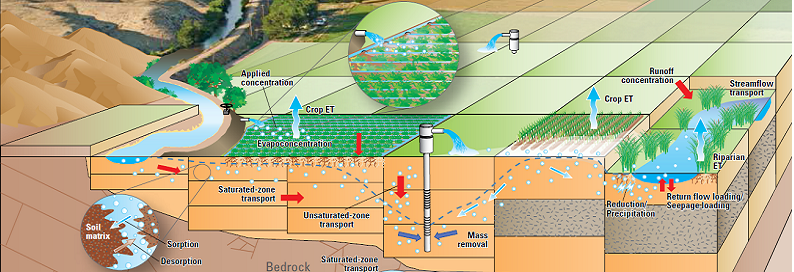



 در منابع آب
در منابع آب




















نظرات (۰)